توضیحات محصول
توالی یابی DNA متیلاسیون | Methylation Sequencing
متیلاسیون سیتوزین ها یک مکانیسم اپی ژنومیک اصلی است که کد ژنومی اولیه را تنظیم میکند. این امر منجر به تشکیل 5-متیل سیتوزینها 5mCs در مکانهای انتخابی ژنوم میشود. متیلاسیون سیتوزین، بیان ژن و بازسازی کروماتین را تنظیم کرده و در نتیجه نقش مهمی در بسیاری از عملکردهای بیولوژیکی از جمله رشد جنینی، تمایز سلولی و پرتوانی سلولهای بنیادی ایفا میکند. متیلاسیون غیر طبیعی DNA می تواند منجر به بیماریهایی مانند سرطان شود.
تعیین توالی متیلاسیون DNA یک فناوری جدیدتر است که معمولاً مبتنی بر تبدیل بیسولفیت برای تمایز سیتوزینهای متیله شده از سیتوزینهای غیرمتیله است. پس از تیمار نمونه با بیسولفیت، سیتوزینهای متیله نشده به اوراسیل تبدیل میشوند، در حالی که 5mCs غیر واکنشی هستند و باقی می مانند. در مرحله تعیین توالی، سیتوزین های متیله نشده به عنوان تیمین خوانده می شوند، در حالی که سیتوزین های متیله هنوز به عنوان سیتوزین خوانده می شوند.
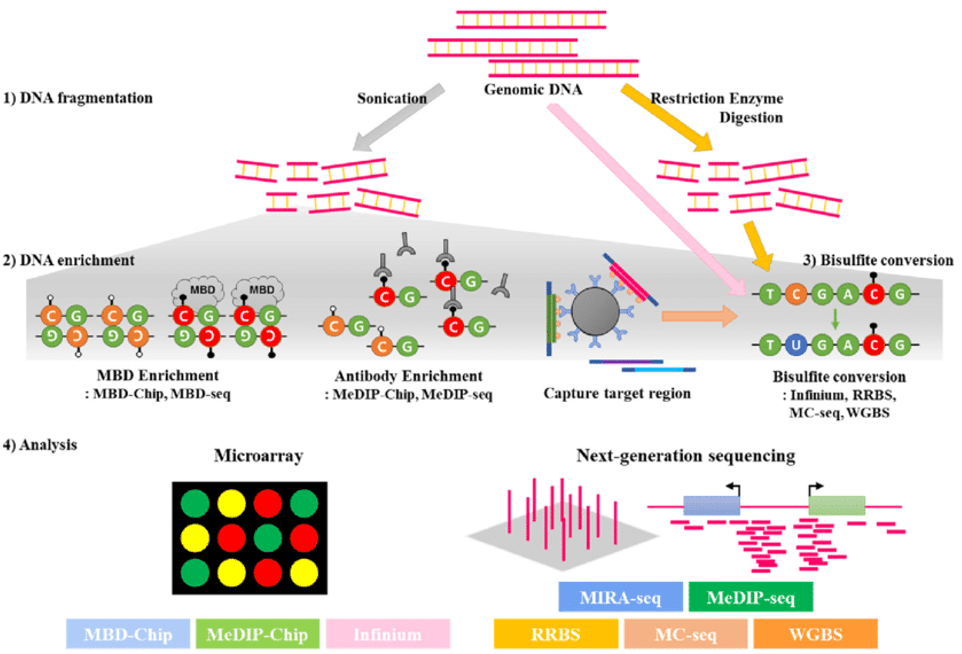
بر اساس پوشش ژنومی، توالی یابی مبتنی بر تبدیل بیسولفیت را می توان در NUSeq به صورت توالی یابی بیسولفیت کل ژنوم یا توالی یابی بیسولفیت (WGBS) با بازنمایی کاهش یافته (RRBS) انجام داد. WGBS هزینه بیشتری دارد و تجزیه و تحلیل داده های مرتبط بسیار بیشتر است. RRBS در عوض یک رویکرد مقرون به صرفه برای بررسی متیلاسیون DNA با نمونه برداری از مناطق غنی از CpG از ژنوم ارائه میدهد. برای انجام RRBS، DNA ژنومی با یک آنزیم محدود کننده حساس به متیلاسیون، مانند MspI هضم می شود. سپس قطعات DNA هضم شده در معرض بستن آداپتور، تبدیل بی سولفیت و PCR قرار می گیرند تا یک کتابخانه برای توالی یابی ایجاد شود.
نحوه عملکرد توالی یابی DNA متیلاسیون | Methylation Sequencing
بسیاری از رویکردها از کیفیت و حساسیت بالای NGS برای تجزیه و تحلیل متیلاسیون استفاده می کنند. اکثر روشها برای شناسایی سیتوزینهای متیله نشده بر تبدیل DNA بی سولفیت متکی هستند. تبدیل بی سولفیت سیتوزین های متیله نشده را در طی آماده سازی کتابخانه به اوراسیل تغییر می دهد. بازهای تبدیل شده بعد از PCR به عنوان تیمین در داده های توالی یابی شناسایی می شوند و از شمارش خوانده شده برای تعیین درصد سیتوزینهای متیله استفاده می شود.توالی یابی تبدیل بیسولفیت را میتوان با روشهای هدفمند مانند آمپلیکون متیل seq یا غنی سازی هدف یا با توالی یابی بیسولفیت کل ژنوم انجام داد.
توالی یابی به روش توالی یابی DNA متیلاسیون | Methylation Sequencing
روش توالی یابی رسوبگذاری ایمنی DNA متیله ( (MeDIP-seqشامل آنتی بادی ضد متیل سیتوزین است. به طور خلاصه، DNA متیله با استفاده از آنتی بادی بر علیه 5-متیل سیتوزین رسوب ایمنی داده می شود و سپس توالی یابی صورت میگیرد. بخشی از DNA که رسوب ایمنی داده میشود، بخش متیله DNA را نشان میدهد و با مقایسه با ژنوم مرجع شناسایی میگردد.
تعیین توالی ژنوم استخراج شده از دامنه متصل به متیل
توالی یابی ژنوم استخراج شده از دامنه متصل به متیل ( (MBDiGs از پروتئینهای نوترکیب MBD و MBD2 برای غنی سازی قطعات DNA غنی از متیل از مخزن DNA ژنومی فراصوت شده استفاده می کند. با توجه به بررسی های انجام شده MBDiGS نسبت به MeDIP-seq ارجحیت دارد زیرا یک گرادیان در غلظت نمک می تواند برای شستشوی قطعات DNA با سرعت های مختلف بسته به وضعیت متیلاسیون آنها استفاده شود.
تعیین توالی آنزیم محدود کننده حساس به متیل
توالی یابی آنزیم محدود کننده حساس به متیل ((MRE-seq، همانطور که از نام آن پیداست، شامل آنزیمهای محدود کننده حساس به متیلاسیون است. نمونه های DNA ژنومی با آنزیمهای محدود کننده هضم میشوند و قطعات DNA بعدی اندازه انتخاب و توالی یابی انجام میگیرد. متیلاسیون DNA افتراقی ممکن است با مقایسه قطعات توالییابی شده بین نمونهها شناسایی شود و اطلاعات مربوط به مکان با مقایسه با ژنوم مرجع شناسایی شود. این روش بخش متفاوتی از ژنوم را در مقایسه با MeDIP-seq تجزیه و تحلیل میکند و بنابراین، میتوان آنها را به عنوان رویکردهای تکمیلی در نظر گرفت.
مزایای توالی یابی DNA متیلاسیون | Methylation Sequencing
متیلاسیون سیتوزین می تواند به طور قابل توجهی بیان ژن زمانی و مکانی و بازسازی کروماتین را تغییر دهد. با بهره گیری از قدرت توالی یابی نسل بعدی NGS هر دو تجزیه و تحلیل گسترده ژنوم و رویکردهای هدفمند می توانند بینشی در مورد الگوهای متیلاسیون در یک سطح نوکلئوتیدی به محققان ارائه دهند.
- الگوهای متیلاسیون نواحی CpG، CHH و CHG را در سراسر ژنوم انسان بررسی میشود.
- مشاهده متیلاسیون عملاً در هر سیتوزین ژنوم در اکثر گونهها با توالییابی بی سولفیت کل ژنوم WGBS، رویکردی در سطح ژنوم.
- تنوع کامل نمونه را با مقادیر کمی DNA ضبط میشود.








ignitonew –
The respective 2D contour plot in Figure 4 shows nadir at the low levels of Nikkol HCO 50 and Lutrol F127