توضیحات محصول
توالی یابی آر ان ای ریبوزومی ۱۶ اس | 16S ribosomal RNA Sequencing
توالییابی 16S rRNA
16S rRNA چیست؟
RNA ریبوزومی S16 (16S rRNA) جزئی از RNA از زیرواحد 30S یک ریبوزوم پروکاریوتی است که به دنباله Shine-Dalgarno متصل می شود و بیشتر ساختار زیر واحد کوچک ریبوزومی ریبونوکلئیک اسید (SSU) را فراهم می کند. ژن های کد کننده آن به عنوان ژن 16S rRNA نامیده می شوند و به دلیل سرعت کم تکامل این ناحیه از ژن، در بازسازی فیلوژنی ها استفاده می شوند. توالی های متعددی از ژن 16S rRNA می تواند در یک باکتری وجود داشته باشد.
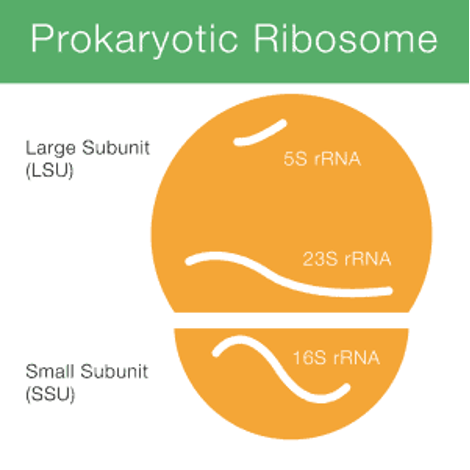
کاربرد توالییابی 16S rRNA
توالی یابی ژن 16S rRNA معمولاً برای شناسایی، طبقه بندی و تعیین کمیت میکروب ها در مخلوط های پیچیده بیولوژیکی مانند نمونه های محیطی (مثل آب دریایی) و نمونههای روده (میکروبیوم روده انسان) استفاده میشود. ژن 16S rRNA÷ یک جزء بسیار حفاظت شده از ماشین رونویسی تمام اشکال حیات مبتنی بر DNA است. بنابراین به عنوان یک ژن هدف برای تعیین توالی DNA در نمونه های حاوی هزاران گونه مختلف بسیار مناسب است. پرایمرهای جهانی PCR را می توان برای هدف قرار دادن مناطق حفاظت شده 16S طراحی کرد و امکان تکثیر ژن را در طیف وسیعی از میکروارگانیسم های مختلف از یک نمونه واحد فراهم کرد.
ژن 16S rRNA از هر دو ناحیه حفاظت شده و متغیر تشکیل شده است. در حالی که منطقه حفاظت شده توسعه جهانی را امکان پذیر می کند، توالی نواحی متغیر امکان تمایز بین میکروارگانیسم های مختلف خاص مانند باکتری ها، آرکیا و یوکاریای میکروبی را فراهم می کند. شناسایی ویروس ها به دلیل نداشتن ژن نشانگر فیلوژنتیکی 16S نیازمند تعیین توالی متاژنومی (توالی یابی مستقیم DNA کل استخراج شده از یک جامعه میکروبی) است.
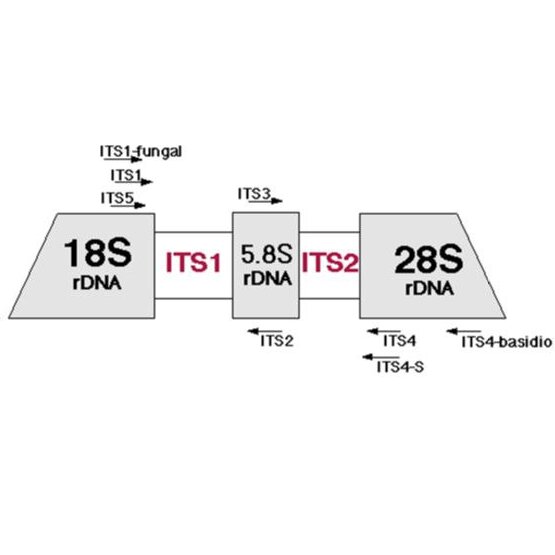
تنوع توالی بین 16S باکتریایی به طور یکنواخت توزیع نشده است. 9 ناحیه بیش متغیر در بین باکتری ها شناسایی شد که V1 تا V9 نامیده می شوند.
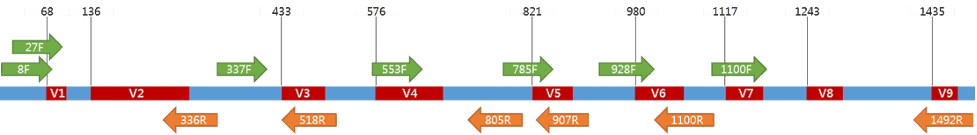
ژن 16S با طول کامل معمولاً توسط جفت آغازگرهای 27F و 1492R و به دنبال آن توالییابی DNA Sanger تقویت میشود. برای به دست آوردن توالی دقیق، هر دو رشته DNA باید با استفاده از پرایمرهای متعدد ارائه شده در جدول زیر توالی یابی شوند.
|
Name of primer |
Sequence |
|
8F |
AGAGTTTGATCCTGGCTCAG |
|
27F |
AGAGTTTGATCMTGGCTCAG |
|
336R |
ACTGCTGCSYCCCGTAGGAGTCT |
|
337F |
GACTCCTACGGGAGGCWGCAG |
|
337F |
GACTCCTACGGGAGGCWGCAG |
|
341F |
CCTACGGGNGGCWGCAG |
|
515FB |
GTGYCAGCMGCCGCGGTAA |
|
518R |
GTATTACCGCGGCTGCTGG |
|
533F |
GTGCCAGCMGCCGCGGTAA |
|
785F |
GGATTAGATACCCTGGTA |
|
805R |
GACTACHVGGGTATCTAATCC |
|
806RB |
GGACTACNVGGGTWTCTAAT |
|
907R |
CCGTCAATTCCTTTRAGTTT |
|
928F |
TAAAACTYAAAKGAATTGACGGG |
|
1100F |
YAACGAGCGCAACCC |
|
1100R |
GGGTTGCGCTCGTTG |
|
1492R |
CGGTTACCTTGTTACGACTT |
تشخیص ژن – 16S rRNA
تکنیک تشخیص ژن 16S rRNA در نتیجه فناوری PCR و پیشرفت های پایدار در فناوری تحقیقات اسید نوکلئیک به پرکاربردترین ابزار برای شناسایی و شناسایی پاتوژن ها تبدیل شده است. این فناوری برای شناسایی، طبقهبندی و یافتن پاتوژنها با سرعت بیشتر و با دقت قابل استفاده است که مراحل آن شامل جمع آوری DNA ژنومی، قطعات ژن 16S rRNA جمع آوری می شود، و توالی ژن 16S rRNA تجزیه و تحلیل می شود.
در اصل، مطالعات نمونههای محیطی نیازمند کشت و جداسازی گونهها برای شناسایی، فرآیندی پرزحمت و زمانبر میباشد. با این حال، جفت شدن 16S rRNA با توالییابی نسل بعدی، مطالعه بسیاری از نمونه ها را با هزینه کم امکان پذیر کرده است. در مطالعات اولیه توالییابی 16S rRNA توالی های زیادی را پیدا شدهاست که به هیچ گونه کشت شناخته شده تعلق ندارند، که نشان می دهد ارگانیسم های غیر جدا شده متعددی وجود دارد و روش های مبتنی بر کشت تنها درصد کمی از گونه های باکتریایی و باستانی را در یک نمونه پیدا می کنند. علاوه بر این، با چندگانه سازی بسیاری از نمونهها و عمق بالای پوشش ارائه شده توسط پلتفرم های نسل بعدی امروزی، اکنون می توانیم نمونه هایی از سری های زمانی جامع را برای کمی سازی پویایی جامعه میکروبی در بسیاری از سایت ها تجزیه و تحلیل کنیم، یا نقشه های سه بعدی دقیقی از جوامع میکروبی تولید کنیم. به عنوان بررسی اینکه آیا تغییرات در گونه های کمیاب یا فراوان با سلامتی و بیماری مرتبط است یا خیر.
خوانشهایی از توالی یابی نسل بعدی را می توان برای شناسایی و طبقه بندی در برابر پایگاه های داده ای مانند پروژه پایگاه داده ریبوزومی (RDP)، GreenGenes و SILVA BLASTED هم تراز کرد. توالیهای مرتبط «خوشهبندی» میشوند و تعداد نمایندگان هر خوشه شمارش میشود. خوشههای توالیهای مشابه به عنوان “واحدهای طبقه بندی عملیاتی” OTUs نامیده میشوند. تعداد OTU در جدولی از فراوانی نسبی برای هر ارگانیسم در هر نمونه خلاصه شده است. تا به امروز، چندین تجزیه و تحلیل برای تجزیه و تحلیل داده های توالی ژن 16S rRNA توسعه داده شده است و دو طبقه بندی رایج مورد استفاده QIIME و Mothur هستند. QIIME کاربران را از خروجی توالی خام خود از طریق آنالیزهای اولیه مانند برداشت OTU، تخصیص طبقه بندی، و ساخت درختان فیلوژنتیک از توالی های نماینده OTU و از طریق تجزیه و تحلیل آماری پایین دستی، تجسم، و تولید گرافیک با کیفیت انتشار میدهد.
تجزیه و تحلیل توالی rRNA 16s
اصل اساسی در تکنیک تجزیه و تحلیل 16S rRNA به دست آوردن اطلاعات توالی 16S rRNA از قطعه ژن در نمونه میکروب از طریق شبیه سازی است. این عمل توسط هیبریداسیون یا برش آنزیم توالی یا پروب و سپس مقایسه آن با دادههای توالی یا اطلاعات مرتبط در اطلاعات 16S rRNA انجام میشود. این کار برای یافتن جایگاه آن در درخت تکاملی و از این رو شناسایی نمونه های احتمالی انجام میگیرد.
توالی یابی RNA ریبوزومی 16S به طور گسترده در میکروبیولوژی برای یافتن تنوع در موجودات پروکاریوتی و سایر موجودات و از این رو مطالعه ارتباط فیلوژنتیکی بین آنها استفاده می شود.
برخی از مزایای استفاده از RNA ریبوزومی در تکنیک های مولکولی عبارتند از:
- RNA ریبوزومی و ریبوزوم ها در تمام سلول ها دیده می شوند
- ژن های RNA حفظ می شوند
- کشت سلول های میکروبی در تکنیک های توالی یابی دیده نمی شود
تعیین توالی ژن rRNA
تعیین توالی ژن rRNA شامل مراحل زیر است:
- جداسازی DNA
- حرارت دادن برای جداسازی رشته ها و پرایمرهای خاص
- طویل سازی پرایمر با DNA پلیمراز
- مراحل بالا را تکرار کنید تا چندین نسخه از ژن 16S rRNA بدست آورید
- انجام الکتروفورز ژل آگارزو بررسی اندازه دقیق محصول
- خالص سازی و تعیین توالی محصول PCR
انواع آنالیز توالی 16S rRNA
قطعه ژن 16S rRNA را می توان در انواع زیر تجزیه و تحلیل کرد:
- هیبریداسیون محصولات PCR با پروب های خاص 16S rRNA برای به دست آوردن اطلاعات ساختار میکروبی. علاوه بر این، کاوشگر را می توان مستقیماً با هیبریداسیون درجا با نمونه آزمایش تشخیص داد.
- محصولات PCR موجود بر روی ناقل پلاسمید توالی یابی شده و با توالی موجود در پایگاه داده 16S rRNA مقایسه می شوند تا جایگاه خود را در درخت تکاملی پیدا کنند و گونه های احتمالی میکروب ها را در نمونه پیدا کنند.
- تجزیه و تحلیل پلی مورفیسم طول قطعه محصولات PCR. نوع ریبوز ژن میکروب با توجه به اطلس الکتروفورز برش آنزیمی و سپس تجزیه و تحلیل عددی و سپس مطالعه با اطلاعات موجود در مجموعه ریبوزوم پیدا میشود. ارتباط بین ساختار میکروبی نمونهها و گونههای میکروبهای مختلف مورد بررسی قرار میگیرد.
کاربردهای 16S rRNA در میکروبیولوژی
- توالییابی ژن 16S rRNA به عنوان روش استاندارد برای شناسایی و طبقه بندی تاکسونومیک گونههای باکتری در نظر گرفته میشود.
- تکنیک های توالی یابی را می توان در توصیف گونه های جدید استفاده کرد، که هرگز با موفقیت در آزمایشگاه ها کشت نشدند.
- می تواند باکتری ها را به جنس ها یا گونه های جدید طبقه بندی کند.
- توالی یابی آن در میکروبیولوژی به عنوان یک جایگزین ارزان و سریع برای تکنیک های فنوتیپی شناسایی باکتریها عمل می کند.
- یک روش ژنتیکی قدرتمند که می تواند منجر به شناسایی پاتوژن های جدید شود.
- برخی از نواحی این توالیهای ژنی، یک توالی امضای خاص گونه را ارائه میدهند که در شناسایی باکتریها استفاده میشود.
- پروبهای نوکلئوتیدی برای شناسایی آنالیز توالی، آنالیز فیلوژنتیک، باکتریهای بالینی و طبقه بندی مولکولی باکتریها استفاده می شوند.








ignitonew –
Been a little chilly but not so bad
بهاره خوش خلق –
سلام ببخشید قیمت توالی یابی 16s را اگر میشه بگین و اینکه به خود پرایمر هم نیاز هست، ممنون
مدیر سایت –
سلام و وقت بخیر
16S (Hiseq PE250)30000 raw tags
85 دلار